| Menu general | ||
| Accueil | ||
| Avant-propos | ||
| Votre avis | ||
| Contacts | ||
| ENZYMOLOGIE | ||
| Quiz 1 | ||
| Quiz 2 | ||
| Quiz 3 | ||
| Quiz 4 | ||
| Quiz 5 | ||
| Quiz 6 | ||
| BIOLOGIE MOLECULAIRE | ||
| Exercice 3 | ||
| Exercice 6 | ||
| Exercice 11 | ||
| BIOINFORMATIQUE | ||
| Exercice 1 | ||
| Exercice 2 | ||
| Exercice 3 | ||
| Exercice 4 | ||
| Exercice 5 | ||
| Exercice 6 | ||
| Exercice 7 | ||
| Exercice 8 | ||
| Exercice 9 | ||
| Exercice 10 | ||
| Exercice 11 | ||
| Exercice 12 | ||
| Exercice 13 | ||
| Exercice 14 | ||
| Exercice 15 | ||
| Exercice 16 | ||
| OUTILS | ||
| Sequence manipulation Suite (SMS) | ||
| Modéliseurs 3D | ||
| ANNUAIRE WEB | ||
| Portails | ||
| Banques nucléiques | ||
| Outils PCR | ||
| Banques protéiques | ||
| Taxonomie | ||
| Bibliographie | ||
| Analyses nucléiques | ||
| Analyses protéiques | ||
| Alignements | ||
| exercice 3 (complet) : La glucose-6-phosphate dEshydrogEnase (G6PD) humaine |
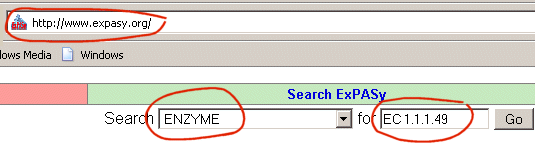
Corrigé 1. Une interrogation de la banque ENZYME du portail EXPASY à l'aide du code de classification
Page d'interrogation de la banque ENZYME du portail EXPASY
Page de résultat : Dans la liste on peut repérer le code de la G6PD humaine (P11413, G6PD_HUMAN)
2. Le fichier de la structure primaire de la protéine (514 acides aminés) peut être récupéré au format FASTA grâce au lien situé en bas à droite de la fiche de l’enzyme. Ce format de fichier comporte un commentaire précédé du symbole > suivi de la séquence en acides aminés codés en lettres avec 80 caractères maximum par ligne.
3. La rubrique 3D structures databases fournit plusieurs liens qui renvoient vers le fichier PDB (code 1QKI). Trois banques proposent des structures 3D (EXPASY, PDB, EBI). Fichier PDB de la structure tridimensionnelle de la G6PD humaine. Il s’agit de la structure d’une variante de l’enzyme mutée (Arg459-Leu) déterminée par cristallographie. Elle se présente sous forme de dimère de dimères. On peut également faire une comparaison dynamique entre monomères.
3. Eléments de synthèse - Déficit en G6PD (favisme) : maladie héréditaire liée au sexe provoquant des anémies hémolytiques. Maladie aggravée par un stress oxydatif (ingestion de fèves) ;
|